Next generation sequencing en non-target screening worden ingezet bij het zoeken naar onbekende micro-organismen (NGS) of onbekende chemische microverontreinigingen (NTS). In dit artikel wordt ingegaan op de overeenkomsten en verschillen en hoe de krachten van deze twee technieken gebundeld kunnen worden.
Geschreven door Nienke Meekel, Aleida Hommes, Michiel Hootsmans, Ton van Leerdam (KWR)
Non-target screening (NTS) en next generation sequencing (NGS) worden allebei ingezet voor geavanceerd speurwerk in de watersector, maar meestal los van elkaar. Tijdens informele discussies tussen microbiologen en chemici bleek al snel dat de twee technieken interessante raakvlakken hebben. Wat zijn de overeenkomsten en de verschillen? Welke uitdagingen zijn er? Kunnen deze technieken elkaar versterken en hoe zou dit eruit kunnen zien? En hoe ga je om met de resultaten en eventuele vertaling naar beleid?
Een korte geschiedenis
Next generation sequencing (NGS)
Sinds het einde van de negentiende eeuw worden bacteriën onder een microscoop of door groei op agarplaten onderzocht en geïdentificeerd op basis van fysiologische en biochemische eigenschappen. Daarmee werd bijvoorbeeld duidelijk hoe ze eruitzien onder een microscoop of wat voor kolonies ze vormen op een agarplaat, welke stoffen ze kunnen afbreken, hoeveel zout ze kunnen verdragen of bij welke temperatuur ze kunnen groeien. Vele combinaties van groeiomstandigheden hielpen wetenschappers uiteindelijk om onderscheid te maken tussen bacteriesoorten. Toch kennen deze methoden hun beperkingen. Niet alle soorten kunnen zo van elkaar worden onderscheiden en, misschien nog belangrijker, de meeste bacteriën kunnen helemaal niet worden gekweekt in een laboratorium.
De ontdekking van DNA als drager van de code van het leven halverwege de twintigste eeuw heeft de identificatie van bacteriën in een stroomversnelling gebracht. Zoals nu alom bekend is het DNA uniek voor elke soort, maar bevat het ook informatie over de fysiologische en biochemische eigenschappen van een soort. Het kraken van deze code ontsluit daarmee de benodigde informatie om bacteriën accuraat te identificeren tot op soortniveau en hun eigenschappen te achterhalen.
Technologieën om de code van het DNA van bacteriën (en andere organismen) te ontcijferen zijn vanaf de jaren zeventig van de vorige eeuw ontwikkeld en steeds sneller en efficiënter geworden. De introductie van Next Generation Sequencing (NGS), begin deze eeuw, heeft dit proces in een stroomversnelling gebracht (zie afbeelding 1). Waar het eerst jaren duurde om de DNA-code van één soort te analyseren kan dit nu binnen enkele uren.
Ook is het mogelijk het DNA van mengsels van micro-organismen te decoderen. Deze technische ontwikkelingen hebben het onderzoek en de ontwikkelingen op het gebied van microbiologie en biotechnologie enorm versneld. Net zo belangrijk is de aanleg en beschikbaarheid van internationale DNA-databanken en referentiebibliotheken waarmee de verkregen DNA-codes worden vergeleken. Zo is het mogelijk om de DNA-codes te identificeren en daarmee te bepalen van welke bacteriesoort deze afkomstig zijn en wat de fysiologische en biochemische eigenschappen zijn.
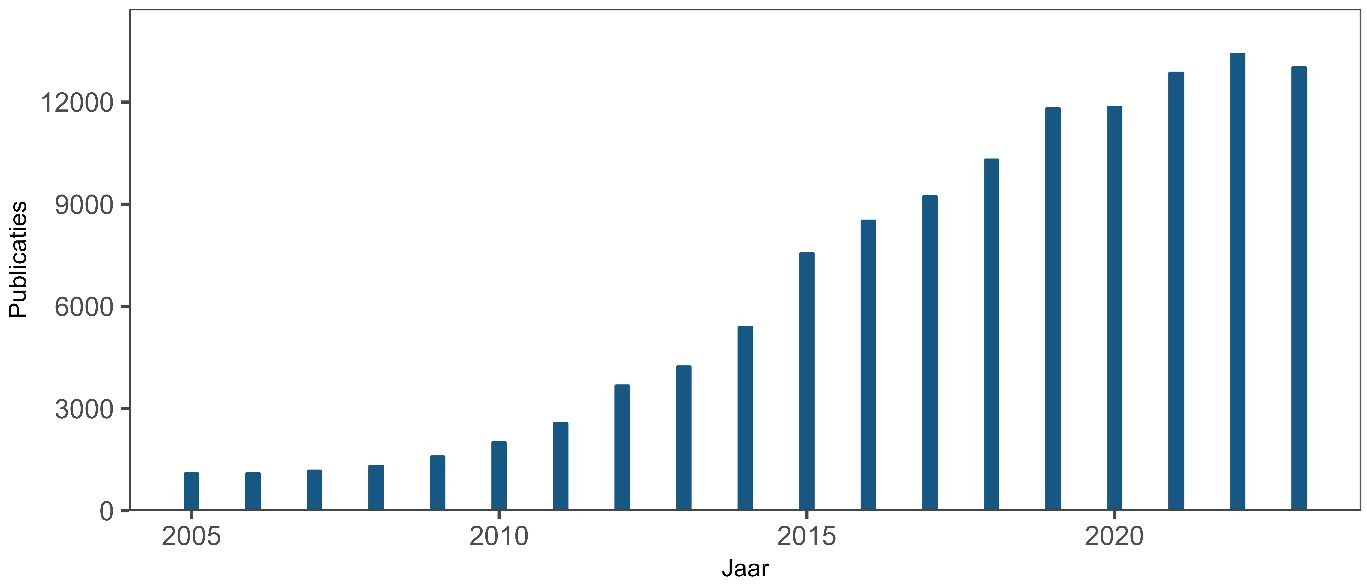
Afbeelding 1. Aantal publicaties in Scopus waarin ‘next-generation sequencing’ of ‘DNA sequencing’ wordt genoemd in de titel, het abstract of de keywords
Tegenwoordig wordt NGS in legio waterkwaliteitsonderzoeken toegepast [1]. Bijvoorbeeld om te beoordelen of er ongewenste bacteriën, zoals pathogenen of bacteriën die putverstoppingen of schade als corrosie kunnen veroorzaken, aanwezig zijn, en/of gewenste bacteriën die bijvoorbeeld verontreinigingen afbreken. De methode wordt ook toegepast om het effect van veranderingen in de omgeving op de samenstelling van micro-organismen te onderzoeken [2]. Voorbeelden zijn hoe een biofilm zich ontwikkelt in de tijd en welk effect temperatuurstijging van het water heeft op de samenstelling van een microbenpopulatie.
NGS wordt ook gebruikt in biodiversiteitsonderzoeken (eDNA) naar bijvoorbeeld vissen of diatomeeën en wordt daarmee ook gebruikt om het herstel (of de afname) van biodiversiteit in watersystemen te monitoren. En er ontstaan via analyse van DNA ook mogelijkheden om vast te stellen wat bacteriën aan het doen zijn: welke biochemische processen voeren ze uit, hoe verandert hun activiteit? Zo kunnen via NGS ook diagnoses worden gesteld over de toestand van een levensgemeenschap en over de effecten van beheer.
Non-target screening (NTS)
De zoektocht naar nieuwe verontreinigingen en onbekende stoffen is een belangrijk onderdeel van het onderzoek naar waterkwaliteit. Dit verkenningswerk speelt al sinds het begin van de jaren zeventig en werd toen vooral uitgevoerd met gaschromatografie en massaspectrometrie. Deze combinatietechniek is echter voornamelijk gericht op vluchtige, apolaire stoffen. Voor het meten van polaire, goed in water oplosbare stoffen is vloeistofchromatografie nodig, maar deze techniek is lastiger toe te passen in de speurtocht naar onbekende stoffen.
De komst van hogeresolutie-massaspectrometers (HRMS) heeft hier verandering in gebracht. De uitvinding van de Orbitrap-analysator en het proof-of-principle door Makarov [3] aan het einde van de jaren negentig was het begin van een reeks technologische verbeteringen die resulteerde in de commerciële introductie van HRMS. De combinatie met de ontwikkeling van milde ionisatietechnieken maakte het mogelijk om HRMS te koppelen aan vloeistofchromatografie. Zo werd de techniek ook geschikt voor watermonsters. Sindsdien is de toepassing van non-target screening (NTS) op watermonsters in opkomst (zie afbeelding 2).[4] Met de opkomst van het internet werd het mogelijk om bibliotheken en databases aan te leggen die onderzoekers wereldwijd kunnen raadplegen en uitwisselen.
Inmiddels is NTS niet meer weg te denken uit de wereld van de analytische milieuchemie. Het wordt momenteel veel toegepast om nieuwe stoffen te ontdekken, verschillen tussen (groepen) monsters te meten en opmerkelijke afwijkingen vast te stellen. Voorbeelden zijn trendanalyses om opkomende stoffen in beeld te krijgen, het in kaart brengen van groepen stoffen en het traceren van lozingen. Enkele voorbeelden van huidige toepassingen worden beschreven in [5].
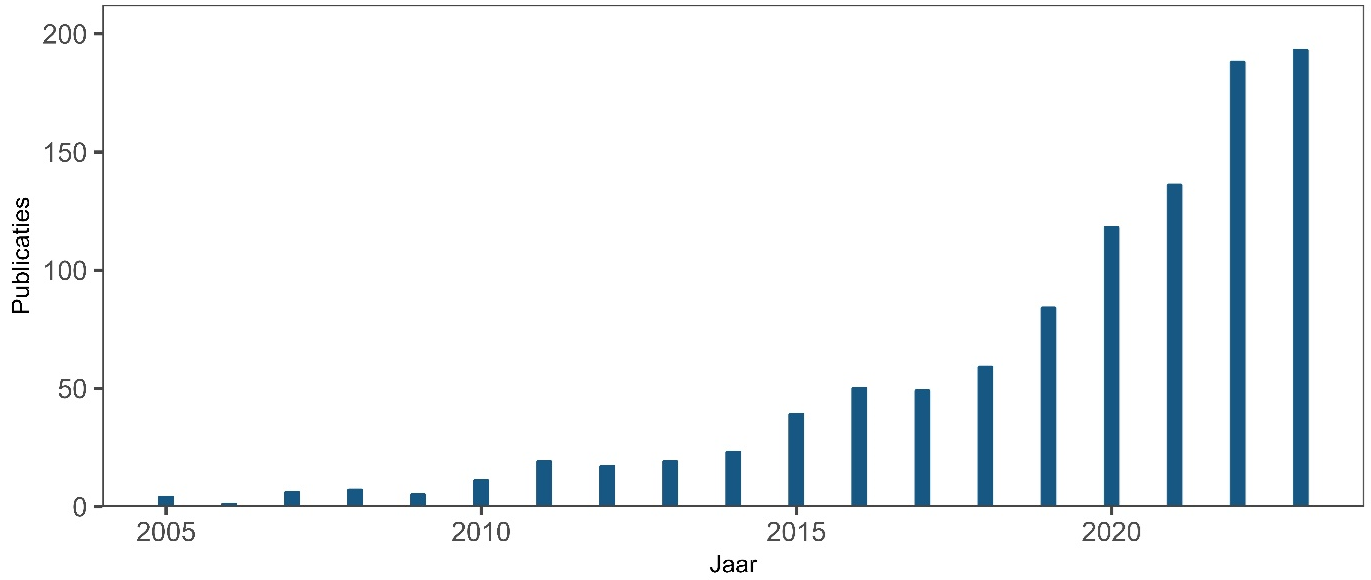
Afbeelding 2. Aantal publicaties in Scopus waarin ‘non-target screening’ of ‘non-target analysis’ wordt genoemd in de titel, het abstract of de keywords
Vergelijkbare workflows en uitdagingen
NGS en NTS worden allebei gebruikt om een zo compleet mogelijk beeld te krijgen van de waterkwaliteit, op microbiologisch of chemisch vlak. Waar normaliter een beperkte set aan specifieke stoffen of micro-organismen gemonitord wordt, geven deze technieken een veel breder beeld en produceren ze een microbiologische of chemische vingerafdruk van een watermonster. Door het generieke karakter van de methodes wordt een grote hoeveelheid stoffen en (micro-)organismen gedetecteerd en kunnen veranderingen en verschillen beter getraceerd worden. Dit past dan ook in het concept van risicogestuurd monitoren (RGM) [6], waarbij zo breed mogelijk gescreend wordt. Desalniettemin blijven de (kwantitatieve) metingen van individuele stoffen en organismen ook van belang om bijvoorbeeld geschikte indicatoren routinematig te monitoren.
De verschillende stappen in NTS en NGS om van monster tot data te komen zijn op hoofdlijnen erg vergelijkbaar (zie afbeelding 3 voor de beide workflows). Het is ook mogelijk om retrospectieve analyses uit te voeren, waarbij historische data opnieuw worden geanalyseerd (mits hetzelfde protocol gebruikt is) en vergeleken met de huidige situatie, of waarmee trends in het verleden ontdekt kunnen worden. Met nieuwe kennis kunnen zo reeds vergaarde data opnieuw onderzocht worden met een andere bril.
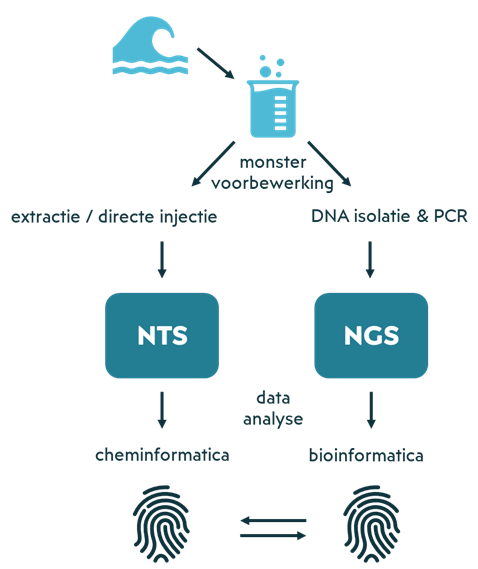
Afbeelding 3. Met non-target screening (links) en next-generation sequencing (rechts) kan een vingerafdruk van het monster bepaald worden. De combinatie van deze vingerafdrukken kan extra informatie opleveren over het monster
Het geheel is meer dan de som van de delen
NTS en NGS geldt zijn beide zichzelf al zeer krachtige technieken waarvan het nut en de toegevoegde waarde al jaren geleden bewezen zijn. Maar tijdens de discussies tussen chemici en microbiologen ontstond al gauw de vraag: wat als deze krachten gebundeld worden?
De kansen liggen in de grootste uitdagingen: NGS en NTS leveren een stortvloed aan data op. Om tot behapbare resultaten te komen is het nodig om keuzes te maken, bepaalde signalen boven andere te prioriteren en ruis uit de data te filteren. Deze keuzes hebben invloed op het eindresultaat: een verkeerde keuze kan leiden tot het missen van belangrijke informatie. Vaak worden hier tools als trend- of timeseries-analyse en clustering op toegepast. Maar wat als de data van beide technieken gebundeld worden? Misschien vertonen bepaalde stoffen eenzelfde trend als bepaalde bacteriesoorten of zijn er tegenovergestelde trends zichtbaar, waarbij bepaalde stoffen afnemen terwijl bepaalde bacteriesoorten tegelijkertijd toenemen.
Zoiets is bijvoorbeeld aangetoond in een onderzoek naar de chemische en microbiologische samenstelling van in- en effluent van een zuivering met snelle zandfiltratie [7]. Dergelijke correlaties zouden kunnen duiden op een verband. Door vervolgens gericht de achterliggende mechanismen en processen te onderzoeken, is te achterhalen of deze verbanden ook oorzakelijk zijn.
Duidelijk is dat deze ontwikkelingen een enorme maar mooie uitdaging opleveren. De data-analyse moet op de vraag ingericht worden om te voorkomen dat men door de bomen het bos niet meer ziet. Datawetenschap speelt hierin ook een belangrijke rol. De benodigde input van verschillende disciplines maakt dat deze vraagstukken op interdisciplinaire wijze beantwoord moeten worden. Omgaan met ‘big data’-analysetechnieken, zoals machine learning en deep learning, helpen om belangrijke informatie te destilleren en de vertaling van deze resultaten naar antwoorden en toepassingen is onderdeel van de werkzaamheden en onderzoeken die KWR uitvoert. Tevens kunnen ontdekte patronen en correlaties leiden tot nieuwe hypotheses over de achterliggende processen of mechanismen.
De kracht van combinatie
De uitdagingen in de toepassing van NGS en NTS lagen in eerste instantie in de technologieontwikkelingen om de brede screening van micro-organismen en chemische componenten mogelijk te maken. Inmiddels zijn de voornaamste uitdagingen het slim en efficiënt omgaan met deze data om (bijvoorbeeld) de waterkwaliteitsvraagstukken van vandaag en de toekomst te helpen beantwoorden en het vertalen van deze data naar bruikbare informatie die in de praktijk ook daadwerkelijk impact heeft.
De ambitie is om NTS en NGS te combineren en zo nog dieper te kunnen delven in de onzichtbare chemische verbindingen en microbiologische soorten in het water. Niet alleen met gegevens uit nieuwe onderzoeken, maar ook terugkijkend naar NGS- en NTS-data die al beschikbaar zijn. Bijvoorbeeld om inzichtelijk te maken welke microverontreinigingen in een zuiveringsstap worden verwijderd en welke micro-organismen daarbij mogelijk zijn betrokken (afbraak) of juist verdwijnen (mogelijk effect van toxiciteit). Gaan sommige chemische en microbiologische processen hand in hand?
Dit komt binnen handbereik door nauwere samenwerking tussen de verschillende vakgebieden en inzet van datawetenschap en/of voorspellende modellering met behulp van kunstmatige intelligentie (AI).
Samenvatting
Twee nieuwe technieken in de moleculaire biologie en analytische chemie zorgen voor grote veranderingen in detectiemogelijkheden: next generation sequencing (NGS) en non-target screening (NTS). Het zijn twee zeer uiteenlopende technieken die worden ingezet bij het zoeken naar onbekende micro-organismen (NGS) of onbekende chemische microverontreinigingen (NTS). In dit artikel wordt ingegaan op de overeenkomsten en verschillen en hoe de krachten van deze twee technieken gebundeld kunnen worden.
REFERENTIES
1. Garner, E. et al (2021). ‘Next generation sequencing approaches to evaluate water and wastewater quality’. Water Research, 194, 116907.
2. Vavourakis, C.D. et al. (2020). ‘Spatial and Temporal Dynamics in Attached and Suspended Bacterial Communities in Three Drinking Water Distribution Systems with Variable Biological Stability’. Environmental Science & Technology, 54, 22, 14535-14546.
3. Makarov, A (2000). ‘Electrostatic Axially Harmonic Orbital Trapping: A High-Performance Technique of Mass Analysis’. Analytical Chemistry, 72, 6, 1156-1162.
4. Hollender, J., Schymanski, E.L., Singer, H.P., Ferguson, P.L. (2017). ‘Nontarget Screening with High Resolution Mass Spectrometry in the Environment: Ready to Go?’ Environmental Science & Technology, 51, 20, 11505-11512.
5. Meekel, N., Leerdam, T. van, Vughs, D., Béen, F., Kotte, M. (2023). ‘Suspect en non-target screening, wat is het verschil?’. H2O-online, 2 juni 2023. https://www.h2owaternetwerk.nl/h2o-podium/uitgelicht/suspect-en-non-target-screening-wat-is-het-verschil, geraadpleegd op 19 maart 2024.
6. Driezum, I. van, Dik, H., Kools, S. (2024) ‘Risicogestuurd monitoren van drinkwaterkwaliteit’ H2O-online, 28 mei 2023. https://www.h2owaternetwerk.nl/vakartikelen/risicogestuurd-monitoren-van-drinkwaterkwaliteit, geraadpleegd op 30 mei 2024.
7. Di Marcantonio, C. et al. (2020). ‘Organic micropollutant removal in full-scale rapid sand filters used for drinking water treatment in The Netherlands and Belgium’. Chemosphere, 260, 127630.


















