Het Vlaamse waterbedrijf Pidpa heeft samen met met TAAG Genetics - Kreglinger een multiplex qPCR-kit ontwikkeld die meerdere indicatoren voor drinkwaterkwaliteit kan detecteren. Na een aanrijkingsstap van slechts 15 uur kan deze methode E. coli, enterococcen en thermotolerante coliformen (genera Citrobacter, Klebsiella, Enterobacter en Escherichia) aantonen.
Download hier de pdf van dit artikel
Geschreven door Katrien de Maeyer (Pidpa)
In samenwerking met TAAG Genetics - Kreglinger heeft het Vlaamse drinkwaterbedrijf Pidpa een qPCR (real-time Polymerase Chain Reaction)-test ontwikkeld. Hiermee kunnen de voornaamste indicatoren van de bacteriologische drinkwaterkwaliteit worden aangetoond in minder dan 24 uur. Hoewel PCR-methoden in de voedingssector al lang ingeburgerd zijn, is de uitdaging om deze techniek in te zetten voor drinkwater net iets groter.
In een omgeving rijk aan voedingstoffen gaan bacteriën zich immers sneller vermenigvuldigen, waardoor hun aanwezigheid makkelijker te detecteren is. In het nutriëntarme milieu dat drinkwater is, gaan bacteriën zich echter niet zo gauw vermeerderen, maar moet alsnog één ongewenste bacteriecel kunnen worden aangetoond. Alle klassieke cultuurmethoden zijn gebaseerd op een kweekstap. Daarbij krijgt het aan te tonen organisme de kans om aan te groeien en is het als gevolg detecteerbaar op of in een geschikt voedingsmedium.
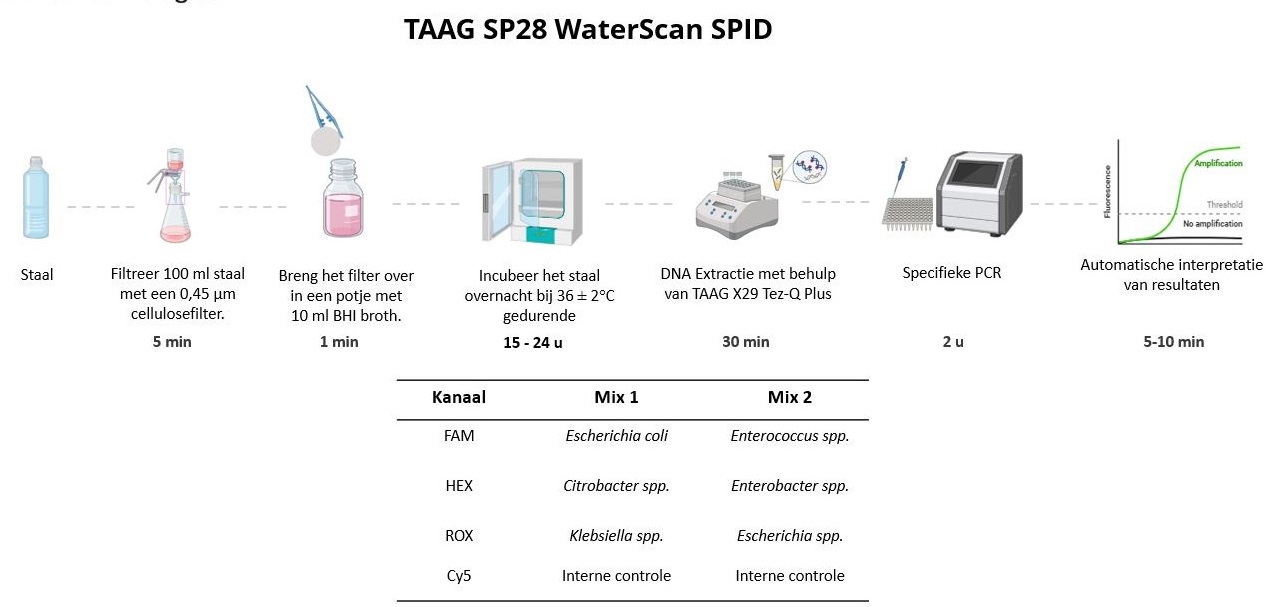
De ontwikkelde techniek combineert de kracht van een kweekstap met de specificiteit van een multiplex qPCR-methode voor verschillende targets. Eerst vindt membraanfiltratie van het drinkwaterstaal plaats, waarna de micro-organismen op het filter minimaal 15 uur kunnen aangroeien in een algemeen vloeibaar medium. Vervolgens wordt op deze aangerijkte cultuur een DNA-extractie (TAAG X29 Tez-Q Plus) uitgevoerd, waarna ten slotte de multiplex qPCR-methode (TAAG SP28 WaterScan SPID) wordt ingezet op het geëxtraheerde DNA (zie afbeelding 1).
De doelorganismen van de qPCR-methode zijn E. coli, de groep van enterococcen (genus Enterococcus) en de thermotolerante coliformen (genera Citrobacter, Klebsiella, Enterobacter en Escherichia). De organismen in deze groep spelen op het gebied van drinkwaterkwaliteit de hoofdrol als indicatoren van fecale verontreiniging en hebben dan ook een grote waarschuwende waarde waarop te ondernemen acties afgestemd worden.
Hoewel de drinkwaterwetgeving voorschrijft om naar de groep van ‘totale coliformen’ te kijken, brengt de methode van TAAG enkel de ‘thermotolerante coliformen’ in beeld. Dit zijn de vier bacteriegeslachten die consequent en met hoge snelheid de biochemische activiteit (lactosevergisting) tot uiting brengen die typerend is voor coliformen en de basis vormt van de huidige onderzoeksmethoden via kweek (ISO 9308 [1], [2]). Aangezien ze bovendien tolerant zijn voor hogere temperaturen, worden ze thermotolerante coliformen genoemd [3]. De overige genera in de coliformgroep vertonen gradaties in de mate waarin ze consequent deze typerende biochemische activiteit uitdrukken.
Bij het ontwikkelen van een PCR-methode voor coliformen is het uitgangspunt eerder de taxonomie dan de biochemie van de organismen. De ‘totale coliformen’ vormen echter geen taxonomisch afgebakende groep, wat de ontwikkeling van een geschikte PCR-methode extra uitdagend maakt. Vandaar dat in de eerste fase van het ontwikkelproces de keuze werd gemaakt om de aandacht te richten op de thermotolerante coliformen, die wel een duidelijk afgebakende groep vormen. Bovendien maken deze reeds een aanzienlijk deel uit van de coliformsoorten die hoger scoren in risicoanalyses (op basis van literatuur, inventarisatie en proceskennis bij het drinkwaterbedrijf). De totale doelgroep van de TAAG-methode (E. coli, enterococcen, thermotolerante coliformen) vormt met andere woorden een mooi uitgangspunt om het potentieel van deze gezamenlijke PCR-ontwikkeling te valideren.
Afbeelding 1. Overzicht van de stappen in het analyseproces
Ontwerp van de test
Aangezien in de PCR-test meerdere doelen bij detectie een ander signaal moeten geven, is een specifieke multiplex-PCR-test ontwikkeld. De grootste uitdaging hierbij was het selecteren van specifieke DNA-segmenten die dienen als startpunten voor het kopiëren en identificeren van het doel-DNA in een PCR-experiment. Hoe goed de PCR-test werkt, hangt af van hoe goed deze DNA-fragmenten zijn gekozen. Als ze goed zijn, kunnen ze alleen de specifieke bacteriestam detecteren.
Het doel van deze multiplex-test is om niet één maar meerdere bacteriesoorten tegelijk op te sporen, waardoor in het PCR-mengsel meerdere van deze DNA-stukjes gecombineerd moeten worden. Dit is riskant, omdat er geen ongewenste soorten gemeld moeten worden en omdat het ook de efficiëntie van de test kan verminderen.
Daarom heeft TAAG Technologies hun op AI gebaseerde multiplex-PCR-ontwerpplatform Mila aan het werk gezet (afbeelding 2). Met gebruik van next-generation machine learning en diverse simulatiemogelijkheden, leverde Mila in korte tijd het beste ontwerp, een proces van trial & error-testen dat anders maanden kan duren. Na een eerste reeks verkennende testen bij TAAG Genetics, werd de PCR-kit ter beschikking gesteld voor validatie bij Pidpa.
Afbeelding 2. Mila als ondersteunend platform om het best mogelijke multiplex-PCR-assay te ontwerpen, met gebruik van met AI opgebouwde kennis in PCR-design
In house validatie
Om de methode te valideren is een vergelijkende studie (conform ISO 16140-2 [4]) uitgevoerd met de referentiemethoden op basis van kweek (ISO 9308 [1], [2]; ISO 7899-2 [5]). Door de verschillende doelstammen in de WaterScan, werd dit een behoorlijk uitgebreide validatie. Per doelsoort werden de inclusiviteit en exclusiviteit bepaald (d.w.z. soorten die respectievelijk wel en niet gedetecteerd worden), alsook de relatieve detectielimiet (relative limit of detection; RLOD) in drinkwater. Daarnaast werd de sensitiviteit bepaald in routinestalen van het distributienetwerk van Pidpa. Om voldoende positieve stalen te kunnen insluiten in de studie, werden ook nog drinkwaterstalen gedopeerd met bacteriologisch meer beladen watermonsters (oppervlaktewater en afvalwater) om verontreinigingssituaties na te bootsen.
Uit deze inclusiviteits- en exclusiviteitstesten bleek dat de methode succesvol de doelstammen kan aantonen en daarnaast ook voldoende specifiek is en dus geen non-targetstammen detecteert. Voor de bepaling van de RLOD werden enkele specifieke doelstammen geselecteerd. Voor alle geteste micro-organismen was de RLOD kleiner dan of gelijk aan 1,0. Dit toont aan dat de ontwikkelde methode, met filtratie, aanrijking en qPCR, deze organismen kan aantonen in waterstalen met minstens een gelijkwaardige gevoeligheid als de referentiemethode (kweekmethode). Tenslotte tonen de resultaten aan dat de methode E. coli, Enterococcus spp., Citrobacter spp., Enterobacter spp., Klebsiella spp. en Escherichia spp. kan aantonen in drinkwaterstalen met een vergelijkbare gevoeligheid als die van de standaard cultuurmethoden, overeenkomstig de aanvaardbaarheidscriteria in ISO 16140-2:2016 [4].
De volledige validatie is in slechts twee weken tijd in house uitgevoerd bij Pidpa. Niet alleen is dit een behoorlijke mijlpaal, het levert ook een intern validatiedossier én opgeleid personeel op. Pidpa trof de nodige voorbereidingen, zoals het verzamelen van relevante stammen (inclusiviteit/exclusiviteit) en stalen (sensitiviteitsstudie) die cruciaal zijn voor de validatie. In de begeleiding van het validatietraject kon Pidpa fulltime rekenen op de expertise van een medewerker van TAAG Genetics. In deze vruchtbare samenwerking konden beide partijen de focus leggen op datgene waar zij het sterkst in zijn. De output, namelijk het validatiedossier, is essentieel voor Pidpa om de methode te implementeren, maar ook waardevol voor TAAG Genetics om de kit op de markt te brengen in de drinkwatersector.
Implementatie bij het drinkwaterbedrijf
Pidpa wil deze methode implementeren in de monitoringsroutine. Ook al levert deze techniek enkel een kwalitatief resultaat op (aanwezig/afwezig) voor de verschillende doelstammen, voor bepaalde monstertypes die niet onder het wettelijk controleprogramma vallen hoeft dit geen probleem te vormen. Daarvoor is vooral de snelheid van het resultaat zeer belangrijk, om zo snel mogelijk de juiste acties te kunnen ondernemen. En in snelheid wint deze techniek, met resultaten in minder dan 24 uur, duidelijk van de kweekmethoden, waarvoor 48 uur nodig is voor enterococcen en coliformen, de nodige tijd voor identificatie tot op soortniveau meegerekend.
Een ander pluspunt is de automatische berichtgeving van de resultaten via de TxA-server, wat erg handig kan zijn bij stalen die buiten diensturen worden aangeleverd. Zo gaat er geen tijd verloren aan interpretatie en rapportage, maar krijgt de betrokken dienst meteen het resultaat in de mailbox.
De manier van werken is evenwel nieuw voor Pidpa, dus in de eerste fase zal de TAAG SP28 WaterScan SPID ‘schaduwdraaien’ naast de gebruikelijke kweekmethoden. Na een gunstige evaluatie kan de kweekmethode voor enterococcen alvast achterwege gelaten worden. De kweekmethode voor coliformen zal vooralsnog worden behouden, aangezien het wenselijk is om de ‘totale coliformen’ in beeld te houden. Naast hun rol als fecale indicator, hebben zij immers ook een relevante rol als procesindicator en bieden de coliform-genera buiten de scope van de WaterScan nog waardevolle informatie.
Het lab (afbeelding 3) is alvast enthousiast over de relatief korte hands-on-time die nodig is voor deze methode, in vergelijking met sommige andere moleculaire methoden. De DNA-extractie bestaat uit slechts enkele stappen en vervolgens kan het DNA meteen geladen worden in ready-to-load PCR-strips, wat zorgt voor een snelle qPCR-reactie. Een niet onbelangrijk detail is dat de prijs van de kit bovendien kan concurreren met de opgetelde prijzen van de kweekmethoden.
Hiernaast gelooft Pidpa sterk in de recent ontwikkelde RT-qPCR-methoden (reverse transcriptase qPCR) voor E. coli en enterococcen, waarbij 1 kolonievormende eenheid (kve) gedetecteerd kan worden in een waterstaal zonder aanrijkingsstap. Deze methode is gevalideerd (in samenwerking met KWR, Vitens, Aqualab-Zuid, Het Waterlaboratorium, WLN en De Watergroep) en de laboranten hebben de techniek ook in de vingers, maar deze is op dit moment voor het Pidpa-lab nog te arbeidsintensief om te implementeren zonder robots of automatisering. Vandaar dat de keuze werd gemaakt om alvast meer in te zetten op de multiplex qPCR met aanrijkingsstap, omdat deze minder arbeidsintensief is, toch ook al 24 uur tijdswinst oplevert en bovendien reeds een brug vormt richting PCR-detectie van de groep van coliformen.
Bij calamiteiten of zeer kritische situaties heeft de RT-qPCR methode wel als troef dat deze zeer snel resultaat oplevert (< 4u) en wordt ook zeker de afweging gemaakt om deze methode in te zetten.
 Afbeelding 3. De validatie werd in het lab van Pidpa uitgevoerd in samenwerking met medewerkers van TAAG Genetics – Kreglinger
Afbeelding 3. De validatie werd in het lab van Pidpa uitgevoerd in samenwerking met medewerkers van TAAG Genetics – Kreglinger
Conclusie en toekomst
TAAG is voor Pidpa een nuttige partner gebleken om een stap vooruit te zetten in moleculaire technieken. Zij beschikken over de nodige expertise om de testkit te ontwikkelen, waar Pidpa de focus kan houden op de praktijktoepassing. Op die manier weet iedereen zeker dat in het validatieonderzoek de meest relevante stammen en geschikte praktijkstalen onderzocht worden. Dit geldt bijvoorbeeld na ingrepen in het distributienet, maar ook voor drinkwaterstalen die gedopeerd werden met een lage dosis oppervlaktewater of afvalwater om een verontreiniging te simuleren. Drinkwaterbedrijven zitten immers bij de bron van een heel spectrum aan waterstalen en bijhorende microbiologische praktijkkennis en geïsoleerde stammen, een waardevol goed dat niet altijd beschikbaar is voor de ontwikkelaars van testkits. Dankzij deze samenwerking kon in een zeer korte doorlooptijd van slechts enkele maanden voor ontwikkeling en validatie, de implementatie van start gaan van een veelbelovende methode.
Ook al detecteert deze methode nog niet alle organismen waarvan dat wenselijk is (bv. totale coliformen), het ontwikkelingsproces legt alvast de brug naar moleculaire technieken. In de toekomst kan er nog steeds gekeken worden naar een uitbreiding of aanpassing van deze PCR-methode, als de verdere evaluatie gunstig verloopt.
Daarnaast zijn er nog samenwerkingsmogelijkheden op het vlak van monitoring van specifieke bedrijfsgerelateerde kwesties, vertaald in een daarvoor op maat ontwikkelde PCR-kit. Deze custom-based manier van werken kan het gebruik van moleculaire technieken in de dagelijkse praktijk in een stroomversnelling brengen.
Samenvatting
Pidpa heeft samen met met TAAG Genetics - Kreglinger een multiplex qPCR-kit ontwikkeld die meerdere indicatoren voor drinkwaterkwaliteit kan detecteren. Na een aanrijkingsstap van slechts 15 uur kan deze methode E. coli, enterococcen en thermotolerante coliformen (genera Citrobacter, Klebsiella, Enterobacter en Escherichia) aantonen. De kit is gezamenlijk ontworpen en gevalideerd, waarbij het samenbrengen van ieders expertise een grote troef bleek voor de voortgang van dit project. Voor Pidpa slaat deze multiplex qPCR een brug naar verdere implementatie van moleculaire methoden, die bovendien de potentie aantoont van verdere gezamenlijke toekomstige ontwikkelingen.
REFERENTIES
1. Nederlands Normalisatie-Instituut (2014). NEN-EN-ISO 9308-1/A1:2017 Water - Telling van Escherichia coli en bacteriën van de coligroep - Deel 1: Methode met membraanfiltratie voor water met een lage achtergrondconcentratie aan bacteriën. NNI, Delft.
2. Nederlands Normalisatie-Instituut (2014). NEN-EN-ISO 9308-2 Water - Telling van Escherichia coli en bacteriën van de coligroep - Deel 2: "Meest waarschijnlijke aantal" methode. NNI, Delft.
3. Baylis, C., Uyttendaele, M., Joosten, H., & Davies A. (2011). The Enterobacteriaceae and their significance to the food industry. ILSI Europe Report Series, Brussels. https://ilsi.eu/wp-content/uploads/sites/3/2016/06/EP-Enterobacteriaceae.pdf, geraadpleegd op 10 september 2024.
4. Nederlands Normalisatie-Instituut (2016). NEN-EN-ISO 16140-2 Microbiologie van de voedselketen - Validatie van methoden - Deel 2: Protocol voor de validatie van alternatieve (eigendomsrechtelijke) methoden tegen een referentiemethode. NNI, Delft.
5. Nederlands Normalisatie-Instituut (2000). NEN-EN-ISO 7899-2 Water - Detectie en telling van enterococcen - Deel 2: Membraanfiltratiemethode. NNI, Delft.