Diatomeeën (kiezelwieren) geven veel informatie over milieu-omstandigheden. Door de afnemende beschikbaarheid van expertise op het gebied van diatomeeën-identificatie is het belangrijk dat de huidige specialisten hun kennis zo efficiënt mogelijk kunnen toepassen en overdragen. Daarbij biedt het voortvarend inzetten van DNA-detectie mooie kansen.
Download hier de pdf van dit artikel
Geschreven door Michiel Hootsmans (KWR Water Research Institute), Kevin Beentjes (Naturalis Biodiversity Center) en Krijn Trimbos (Universiteit Leiden)
Om de ecologische kwaliteit van Europese wateren op het gewenste niveau te krijgen en te houden moeten waterbeheerders monitoring- en maatregelpakketten implementeren. Daarmee dragen ze bij aan het behalen van de doelen van de Kaderrichtlijn Water (KRW). Als maat voor biologische waterkwaliteit worden voor de KRW vier groepen indicatororganismen gebruikt: vissen, macrofauna, water- en oeverplanten en algen [1]. Deze groepen verschillen in hun reactie op veranderingen in hun leefgebied, en in de ruimtelijke omvang daarvan. In samenhang geven ze daarom een completer beeld van de kwaliteit van hun omgeving.
Diatomeeën (kiezelwieren) reageren snel op veranderingen in hun milieu en van veel soorten is bekend onder welke omstandigheden ze voorkomen [2], [3]. Ze worden gebruikt om de waterkwaliteit te bepalen in stromende en stilstaande wateren, maar zijn niet zo eenvoudig op naam te brengen. Dit maakt analyses tijdrovend en daardoor tamelijk kostbaar. Ook kunnen determinatiefouten en -verschillen tussen experts optreden. Daarmee zijn verkeerde conclusies niet uitgesloten.
DNA-detectietechnieken
De genoemde problemen kunnen worden beperkt met nieuwe, op DNA gebaseerde identificatiemethoden [4], [5]. In een zogeheten DNA-metabarcode-analyse kunnen soortspecifieke stukjes DNA van diatomeeën in een monster, barcodes genaamd, geautomatiseerd op naam worden gebracht met een DNA-barcodebibliotheek.
Deze barcodebibliotheek is nu nog niet zo uitgebreid als de voor microscopisch onderzoek beschikbare referentiesoortenlijst. Er zijn echter ook al studies die de aanwezigheid van DNA-barcodes van diatomeeën direct correleren met waterkwaliteitsinformatie zonder dat de barcodes op naam hoeven te worden gebracht [6]. Zo’n alternatieve, taxonomievrije benadering met DNA-barcodes biedt wellicht ook kansen voor een waterkwaliteitsbepaling met DNA. De (nog) beperkte soortendekking van de barcodebibliotheek zou dan geen rol meer spelen, evenmin als mogelijk foute taxonomische identificaties.
Uitdaging en aanpak
In de periode 2020-2024 is het project DNA Diatom Biosensor uitgevoerd [7] met als doel om voor diatomeeën een DNA-detectiemethode te ontwikkelen en daarmee een quickscan voor de waterkwaliteit. Hierbij wordt de ook in Nederland toegepaste IPS-waterkwaliteitsindex (‘Indice de Polluosensitivité Spécifique’ [1]) gebruikt. Deze index weerspiegelt de samenstelling van de diatomeeëngemeenschap in reactie op organische belasting en loopt van 0 (zeer slechte waterkwaliteit) tot 20 (zeer goed). Voor een groot aantal soorten zijn IPS-indicatiewaarden bekend.
De gebruikte monsters voor microscopische identificatie en DNA-analyse kwamen uit reguliere monitoring van de deelnemende waterbeheerders en uit hun pilotstudies naar bijvoorbeeld effecten van aangepast landbouwkundig gebruik en waterbeheer. Naturalis Biodiversity Center voerde de DNA-analyse samen met BaseClear uit, met een zogeheten Illumina Novaseq-sequencer en deels met een Illumina Miseq-sequencer. Beschrijvingen van de werkwijze [8] en de gebruikte DNA-barcodebibliotheek [9] zijn beschikbaar.
Vergelijking van met microscopie en met DNA bepaalde soortenlijsten
De gebruikte, taxonomisch goed geborgde barcodebibliotheek voor diatomeeën beslaat slechts 7 procent van de soorten, ondersoorten en variëteiten die voor microscopische analyse bekend zijn. Ook ligt het aandeel soorten met een ‘goede’ IPS milieu-indicatiewaarde in deze DNA-lijst significant lager dan in de microscopielijst. Dit maakt het risico op een onderschatting van de IPS-indexwaarde met DNA groter dan met microscopie.
De DNA-detectie leverde resultaten op voor 94 procent van de 363 aangeleverde monsters. In de DNA-Novaseq-dataset (35 miljoen sequenties of ‘reads’) werden met de op diatomeeën-DNA gerichte analyse 1.719 unieke barcodes aangetroffen (‘exact sequence variants’, ESVs). Hiervan behoorde 98,5% tot het fylum Bacillariophyta (diatomeeën) en alleen deze zijn verder gebruikt. Dit hoge percentage bewijst dat de DNA-analyse inderdaad heel specifiek was voor het detecteren van diatomeeën-DNA. Van de 1.719 barcodes konden er 532 worden geïdentificeerd tot op het niveau van diatomeeënsoort.
Dit resulteerde in 185 verschillende diatomeeënsoorten (minder dan 532, omdat sommige morfologisch als één soort geïdentificeerde diatomeeën meerdere ESVs hebben). Deze 185 diatomeeënsoorten omvatten 69% van alle 35 miljoen gevonden sequenties. Voor de resterende sequenties (die dus wel voor het overgrote deel als diatomee waren geïdentificeerd) kon met de huidige DNA bibliotheek geen soortnaam worden vastgesteld. Met microscopie werden in dezelfde dataset 492 diatomeeënsoorten vastgesteld.
De soortensamenstelling verschilde dus sterk tussen de analyse met de microscoop en met DNA. De lijst van alle in de totale dataset aangetroffen soorten omvatte in de microscopische analyse ongeveer 2,5 maal (492/185) zoveel soorten als met DNA. Dit is te verklaren door de beperkte dekking van de DNA-referentiebibliotheek. Per monster bleken er echter met DNA twee keer zoveel soorten te worden aangetroffen als op basis van microscopie.
Mogelijke oorzaken voor deze verschillen zijn: het met DNA wel kunnen vaststellen van microscopisch lastig te identificeren (vaak kleine) soorten, het determineren van lege schaaltjes van dode cellen (die met DNA niet worden waargenomen) en foutieve identificaties met de microscoop of DNA. Bovendien speelt waarschijnlijk mee dat het in Nederland gebruikelijk is om per monster 200 diatomeeschaaltjes met de microscoop te beoordelen, terwijl de DNA-analyse gemiddeld 167.000 reads per monster opleverde. Zo’n groot verschil in steekproefgrootte binnen een monster kan leiden tot een ander beeld van het in een monster aanwezige soortenpalet [10].
Vergelijking van de IPS-indexwaarden op grond van microscopie en DNA
Met de DNA- en microscopiedatasets werden voor elk monster resultaten berekend voor de bovengenoemde IPS-index, die loopt van 0 tot 20. Beide datasets waren positief met elkaar gecorreleerd. Er was ook veel variatie, waarbij de IPS-waarden op basis van DNA-gegevens gemiddeld 1,7 IPS-eenheden lager waren dan die op grond van microscopie. Dat zal deels zijn veroorzaakt doordat er in de gebruikte DNA-barcodebibliotheek relatief meer soorten aanwezig waren met een lagere IPS-score.
Uit de wetenschappelijke literatuur bleek dat een bandbreedte van 1 tot 2 (en vaak zelfs 4) IPS-eenheden bij microscopische analyse als normaal kan worden beschouwd, en dat de reproduceerbaarheid van de huidige microscopische methode haar beperkingen kent [11]. De reproduceerbaarheid is met gebruik van DNA-barcodes heel goed en de hier geconstateerde onderschatting van de IPS-indexwaarde met DNA valt binnen de blijkbaar normale bandbreedte van de met microscopie bepaalde IPS.
Met de IPS-index is ook de KRW-waterkwaliteitsklasse voor de hiervoor beschikbare 203 locaties bepaald. Deze indeling bestaat uit vijf klassen, van slecht tot zeer goed, met elk een breedte van 4 IPS-eenheden. De resultaten voor DNA en microscopie waren voor 46 procent van de locaties gelijk (zie afbeelding 1). Voor 33% lagen ze met DNA één klasse lager, voor 11% één klasse hoger.
Deze mate van overeenstemming in KRW-klasse-indeling tussen microscopie en DNA wordt ook elders gevonden: een variatie van 10% in de geschatte waterkwaliteits-indexwaarde is daarbij heel normaal [6], [12]. Voor de IPS komt dat neer op 2 IPS-eenheden, overeenkomend met een halve KRW-kwaliteitsklasse. Daarmee leidt zo’n variatie makkelijk tot een indeling van een monster in een naastgelegen KRW-kwaliteitsklasse.
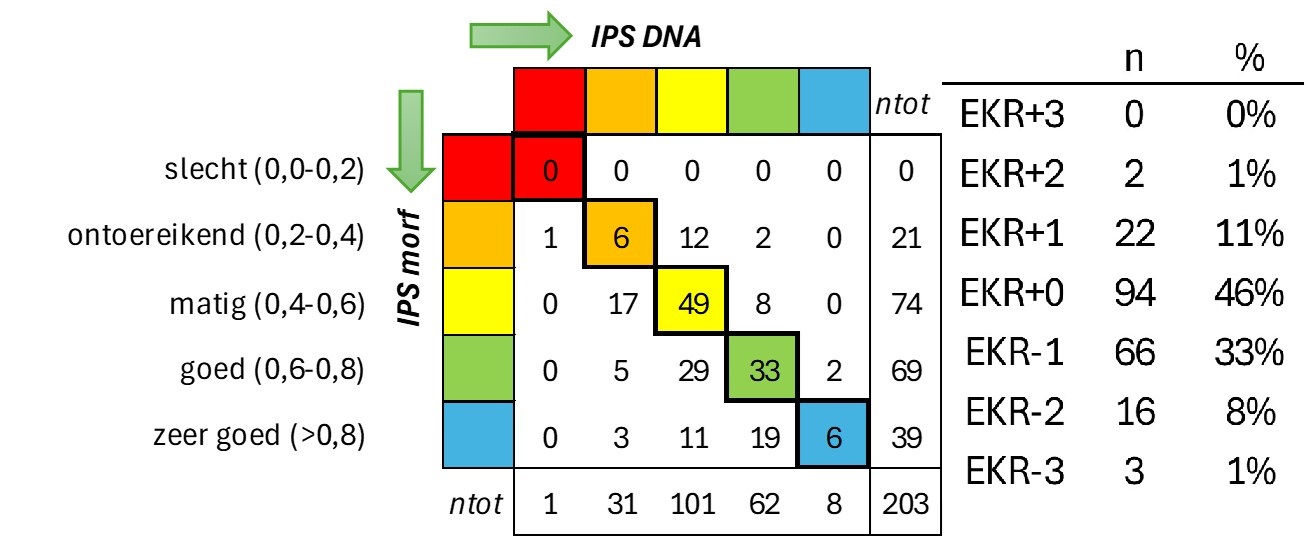
Afbeelding 1. Verdeling van 203 monsters over KRW-kwaliteitsklassen: van boven naar beneden op grond van de IPS bepaald met microscopie (IPS morf), van links naar rechts op basis van de IPS bepaald met DNA. De afwijking tussen de KRW-kwaliteitsklasse volgens DNA en volgens microscopie is samengevat in de rechterhelft van de tabel; bijvoorbeeld: de regel met ‘EKR+1’ betekent dat 22 locaties (11% van het totale aantal) volgens DNA één KRW-kwaliteitsklasse hoger uitkomen dan op grond van microscopie
Taxonomie-vrije KRW-classificatie op basis van de DNA-barcodegemeenschap
De taxonomie-vrije aanpak werd toegepast met een machine learning-techniek (zie [13]) gebaseerd op alleen de DNA-barcodesamenstelling van de daarvoor beschikbare locaties (deze barcodes werden nu dus níet gedetermineerd tot op soort). Met 75 procent van deze locaties werd een model getraind om de KRW-kwaliteitsklassen, bepaald op grond van de microscopische gegevens, te voorspellen met alleen de DNA-barcodesamenstelling van de monsters.
Vervolgens werd dit model getest door de KRW-klasse voor de resterende 25 procent van de locaties te voorspellen op basis van hun DNA-barcodesamenstelling. Dit gaf in 46 procent van de gevallen eenzelfde KRW-klasse als met microscopie was bepaald (zie afbeelding 2). Daarmee is de kwaliteit van deze taxonomie-vrije DNA aanpak om de KRW-klasse te bepalen vergelijkbaar met het resultaat waarbij de uit de DNA-barcodes afgeleide soortenlijsten per locatie werden gebruikt (afbeelding 1).
Als de KRW-kwaliteitsklassen ‘ontoereikend’ en ‘matig’, en de klassen ‘goed’ en ‘zeer goed’ werden samengenomen tot twee klassen die evenveel monsters bevatten, was de juistheid van de machine learning-voorspelling 67%. In 85 procent van de gevallen werd ook het juiste watertype (stilstaand of stromend) correct voorspeld.
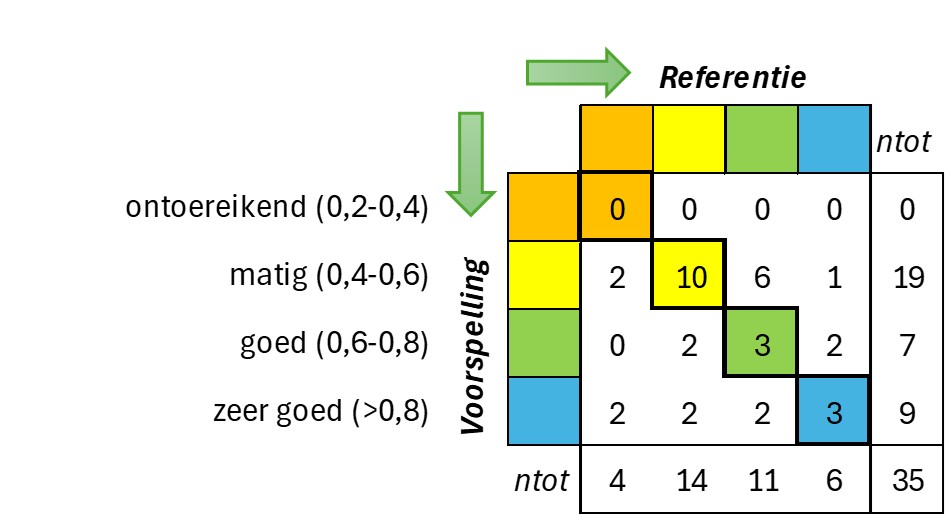
Afbeelding 2. Voorspelling van KRW-kwaliteitsklassen van locaties met behulp van machine learning, op grond van alleen hun DNA-barcodesamenstelling ten opzichte van de bijbehorende microscopisch bepaalde KRW-kwaliteitsklassen (referentie)
De taxonomie-vrije werkwijze met alleen ESVs (afbeelding 2) leverde dus geen beduidend grotere ‘correcte’ voorspelling op van de KRW-kwaliteitsklassen in de dataset, dan de werkwijze met op DNA gebaseerde soortenlijsten (afbeelding 1). Dat werd wel verwacht, doordat in het machine learning-model ook de niet-taxonomisch geïdentificeerde ESVs konden bijdragen aan de typering van een KRW-kwaliteitsklasse (zo’n verbetering werd bijvoorbeeld gevonden in [14]). Wat wél opvalt is dat in vergelijking met afbeelding 1 de verdeling van de ‘verkeerd’ ingedeelde locaties in afbeelding 2 veel meer symmetrisch is rond de diagonaal van ‘correct’ voorspelde kwaliteitsklassen. De aanvullende kwaliteitsinformatie uit de niet tot soort geïdentificeerde ESVs droeg in dit opzicht dus wel degelijk positief bij, naast de informatie die de in principe ook tot soort te identificeren ESVs in het model al inbrachten.
Praktijksituatie op basis van DNA
Ter illustratie zijn diverse praktijksituaties geanalyseerd, waarbij alleen op DNA gebaseerde soortenlijsten beschikbaar waren. Hierbij werden naast de IPS-index ook de Van Dam-indexen [2] bepaald. Deze Van Dam-indexen maken gebruik van de indicatiewaarden van diatomeeën voor zeven milieu-aspecten. Voor de Van Dam-saliniteitsindex zijn bijvoorbeeld vier indicatiewaarden beschikbaar (1 tot en met 4); een soort die voorkomt in zoetwater scoort een 1, een soort uit een vrij brak water een 4. Verder zijn er Van Dam-indexen voor pH (zuurgraad; zes niveaus), stikstofmetabolisme (behoefte aan organisch gebonden stikstof; vier niveaus), zuurstofmetabolisme (gewenste mate van zuurstofverzadiging; vijf niveaus), saprobie (belasting met organische stof; vijf niveaus), trofiegraad (belasting met nutriënten; zeven niveaus) en vochtigheid (indicatie voor de mate waarin diatomeeën droogval verdragen; vijf niveaus).
De waarde van elke Van Dam-index wordt bepaald als de som van de bij die index geldende indicatiewaarde van elk individu in een monster, gedeeld door het totale aantal individuen in het monster. Bij de DNA-gegevens is elke op soortnaam gebrachte barcode beschouwd als een individu. De Van Dam-indexen geven samen een breder palet aan waterkwaliteitsinformatie uit diatomeeën dan alleen de IPS. Afbeelding 3 geeft een voorbeeld van deze aanpak op drie locaties in Landgoed Oostbroek, in het Kromme Rijngebied (provincie Utrecht).
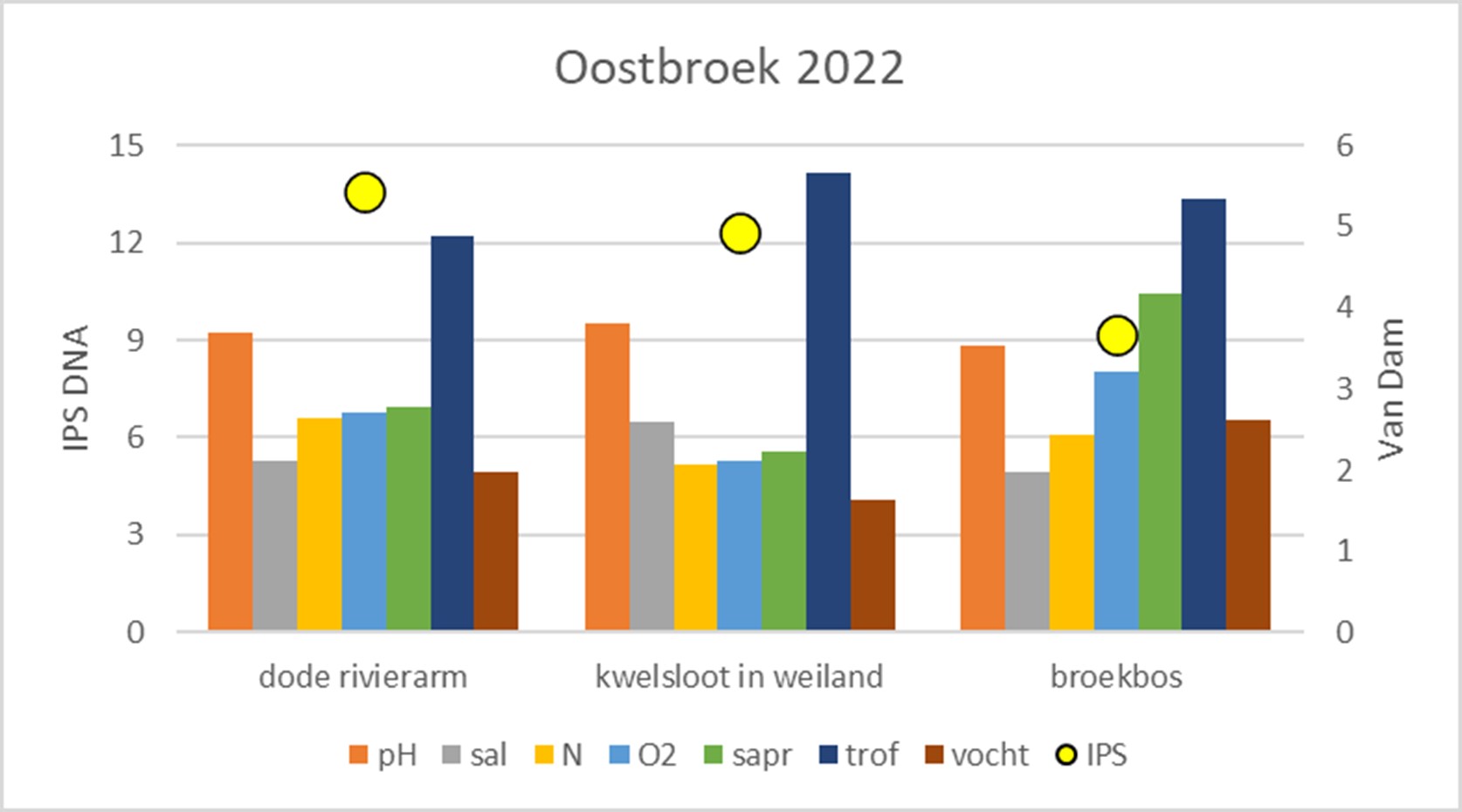
Afbeelding 3. Drie locaties in landgoed Oostbroek, 2022, met voor elk de op DNA-resultaten gebaseerde waarden voor de IPS-index (gele bollen) en de zeven Van Dam-indexen (staafjes: pH=zuurgraad; sal=zoutgehalte; N=organische stikstofbelasting; O2=zuurstofverzadiging; sapr=organische stofbelasting; trof=voedselrijkdom; vocht=indicatie voor mate van droogval)
De relatieve bijdragen van de afzonderlijke Van Dam-indicatiewaarden die samen de gemiddelde waarde bepalen van een Van Dam-index, geven nog meer informatie over de milieu-omstandigheden en de verschillen tussen locaties. Afbeelding 4 geeft hiervan nog twee voorbeelden uit Oostbroek. De Van Dam-index voor organische stikstofbelasting in de rivierarm heeft een sterke bijdrage van soorten met indicatiewaarde 4. Deze soorten hebben een continu verhoogde organische stikstofconcentratie nodig.
In de kwelsloot komen voornamelijk soorten met indicatiewaarde 2 voor, die organische stikstofbelasting wel kunnen verdragen maar het niet nodig hebben. Het broekbos heeft naast dergelijke soorten ook soorten die periodiek organische stikstof nodig hebben (indicatiewaarde 3). De analyse laat een geleidelijke stijging zien in organische stikstofbelasting: laag in de kwelsloot via matig in het broekbos naar hoog in de rivierarm.
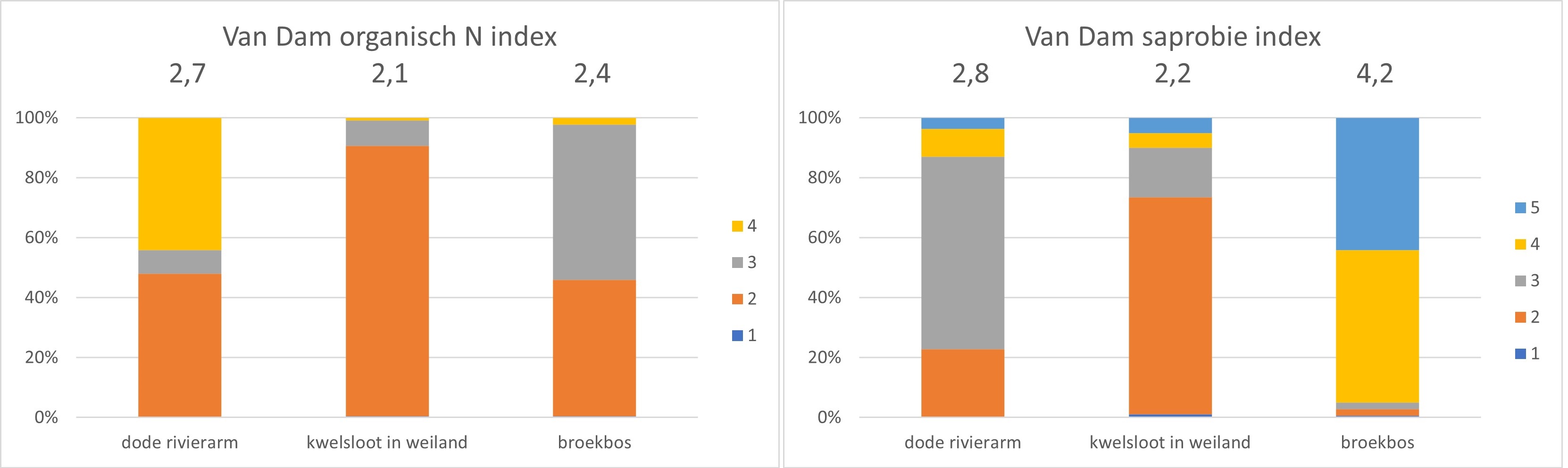
Afbeelding 4. Drie locaties op landgoed Oostbroek, 2022. Relatieve bijdrage van de onderliggende indicatiewaarden aan de Van Dam-indexwaarde voor belasting met organische stikstof (links) en organische stof (rechts). Het getal boven elk staafdiagram is de indexwaarde. Betekenis van de indicatiewaarden: hoger=toename van belasting met organische stikstof respectievelijk organische stof
Conclusies en kansen
Diatomeeën geven veel informatie over aquatische milieu-omstandigheden. Door hun korte levenscyclus zijn het relatief snelle en gevoelige indicatoren ten opzichte van andere organismen, zoals waterplanten en vissen.
Voor acceptatie van DNA-detectieresultaten is het van belang dat deze vergelijkbare kwaliteitsoordelen opleveren ten opzichte van de bekende en vertrouwde methode van microscopie. Dit is in dit onderzoek bevredigend aangetoond, afgezet tegen de normale bandbreedte van microscopie-data. Soms geven DNA-detectie en microscopie een wat ander beeld van de werkelijkheid. Het is echter niet eenvoudig te bepalen welk van beide benaderingen 'goed' of 'beter' is. Beide methoden leiden dan ook niet altijd tot een overeenkomstige waarde voor waterkwaliteitsklassen.
Dit is geen reden om DNA-detectie alleen als pilotinstrument te zien. Ook de aanpak met microscopie geeft aanleiding tot serieuze vragen rond de mate van variatie en daarmee de betrouwbaarheid en reproduceerbaarheid van resultaten. Het is daarom aanbevelenswaardig om naast de microscopische methode ook DNA-detectie in te zetten bij routinematige monitoring en in waterkwaliteitsonderzoek. Zo kan de DNA Diatom Biosensor-methode in zowel stromende als stilstaande wateren verder worden ontwikkeld om te komen tot een betere op diatomeeën gebaseerde diagnose en beoordeling van de waterkwaliteit.
Dankbetuiging
De auteurs bedanken alle partners in het TKI-samenwerkingsproject ‘DNA Diatom Biosensor’ voor hun onmisbare bijdragen: de waterbeheerders Brabantse Delta, Delfland, De Dommel, Fryslân, Hollandse Delta, Hollands Noorderkwartier, Limburg, Rijnland en De Stichtse Rijnlanden; STOWA en BaseClear; de waterlaboratoria Waterproef en AQUON en de betrokken kennisinstellingen.
Financiering voor dit onderzoek kwam mede uit de Toeslag voor Topconsortia voor Kennis en Innovatie (TKI's) van het ministerie van Economische Zaken en Klimaat (Topsector Water).
Samenvatting
Diatomeeën (kiezelwieren) geven veel informatie over milieu-omstandigheden. Met hun korte levenscyclus zijn het snelle en gevoelige indicatoren. Identificatie van onder meer diatomeeën met DNA maakt een fijnmazige monitoring in ruimte en tijd mogelijk die recht doet aan de complexiteit en dynamiek van aquatische ecosystemen. Daarmee kan de kwaliteit van diagnostiek en daarop gebaseerde maatregelen voor inrichting en beheer worden verbeterd. Door de afnemende beschikbaarheid van expertise op het gebied van diatomeeën-identificatie is het belangrijk dat de huidige specialisten hun kennis zo efficiënt mogelijk kunnen toepassen en overdragen. Daarbij biedt het voortvarend inzetten van DNA-detectie mooie kansen.
REFERENTIES
1. Molen, D.T. van der, Pot, R., Evers, C.H.M., Herpen, F.C.J. van & Nieuwerburgh, L.L.J. van (2020). Referenties en maatlatten voor natuurlijke watertypen voor de Kaderrichtlijn Water 2021-2027. Stowa 2018-49, versie juni 2020. https://www.stowa.nl/sites/default/files/assets/PUBLICATIES/Publicaties_2018/STOWA_2018-49-Maatlatten v2024 DEF.pdfhttps://www.stowa.nl/sites/default/files/assets/PUBLICATIES/Publicaties_2018/STOWA_2018-49-Maatlatten v2024 DEF.pdf, geraadpleegd op 17 oktober 2024.
2. Dam, H. van, Mertens, A., & Sinkeldam, J. (1994). ‘A coded checklist and ecological indicator values of freshwater diatoms from The Netherlands’. Neth. J. Aquat. Ecol. 28: 117-133. https://doi.org/10.1007/BF02334251
3. Wal, J. van der, Koning, J. de & Dam, H. van (2024). ‘Snel inzicht in de ecologische waterkwaliteit met diatomeeën’. H2O-Online, 13 juni 2024. https://www.h2owaternetwerk.nl/vakartikelen/snel-inzicht-in-de-ecologische-waterkwaliteit-met-diatomeeen
4. Beentjes, K.K. (2020). DNA-technieken voor waterbeheerders. Stowa Deltafact, https://www.stowa.nl/deltafacts/waterkwaliteit/diversen/dna-technieken-voor-waterbeheerders, geraadpleegd op 17 oktober 2024
5. Trimbos, K., Beentjes, K.K & Hootsmans, M.J.M. (2024). ‘DNA-technieken voor waterbeheerders: stand van zaken en uitdagingen’. H2O-Online, 5 juni 2024.
https://www.h2owaternetwerk.nl/vakartikelen/dna-technieken-voor-waterbeheerders-stand-van-zaken-en-uitdagingen
6. Apothéloz-Perret-Gentil, L., et al. (2017). ‘Taxonomy-free molecular diatom index for high-throughput eDNA biomonitoring’. Mol. Ecol. Res., 17:1231–1242. https://doi.org/10.1111/1755-0998.12668
7. Hootsmans, M.J.M., Beentjes, K.K., Wal, J. van der & Dam, H. van (2024). DNA Diatom Biosensor - Ontsleutelen van milieukwaliteit op basis van diatomeeën. KWR rapport 2024.081. https://www.tkiwatertechnologie.nl/projecten/dna-diatom-biosensor/, geraadpleegd op 17 oktober 2024
8. Beentjes, K.K., (2024). TKI Diatom NGS Protocol. Online op Protocols.io: https://dx.doi.org/10.17504/protocols.io.6qpvr8k4plmk/v1
9. Rimet, F., Gusev, E., et al.. (2019). ‘Diat.barcode, an open-access curated barcode library for diatoms’. Sci. Rep. 9:15116, https://doi.org/10.1038/s41598-019-51500-6, geraadpleegd op 17 oktober 2024.
10. Verweij, G., Wanink, J.H. & Kouwets, F.A.C. (2015). ‘As time goes by - Veranderingen in soortconcepten in relatie tot analyseprotocollen en ecologische beoordelingen’. Diatomededelingen 39: 19-27.
https://www.nvkd-diatom.nl/_files/ugd/f21ff7_5bd8fd29829a45cea8d15f2366de8bf6.pdf#page=19, geraadpleegd op 17 oktober 2024
11. Besse-Lototskaya, A., Verdonschot, P.F.M. & Sinkeldam, J.A., (2006). ‘Uncertainty in diatom assessment: Sampling, identification and counting variation. In: Furse, M.T., Hering, D., Brabec, K., Buffagni, A., Sandin L. & Verdonschot, P.F.M., (eds). ‘The Ecological Status of European Rivers: Evaluation and Intercalibration of Assessment Methods’. Developments in Hydrobiology, vol 188. Springer, Dordrecht. https://doi.org/10.1007/978-1-4020-5493-8_17
12. Kelly, M.G., Juggins, S., et al. (2020). ‘Development of a novel metric for evaluating diatom assemblages in rivers using DNA metabarcoding’. Ecol. Indic. 118: 106725. https://doi.org/10.1016/j.ecolind.2020.106725 .
13. Kuhn, M. (2008). ‘Building predictive models in R using the caret package’. Journal of statistical software, 28, 1-26. https://doi.org/10.18637/jss.v028.i05
14. Gregersen, R. et al.. (2023). ‘A taxonomy-free diatom eDNA-based technique for assessing lake trophic level using lake sediments’. J. Env. Manage. 345: 118885, https://doi.org/10.1016/j.jenvman.2023.118885















